Del ácido desoxirribonucleico (ADN) a las proteínas
| Sitio: | Aulas | Uruguay Educa |
| Curso: | Química - 3º B.D. |
| Libro: | Del ácido desoxirribonucleico (ADN) a las proteínas |
| Imprimido por: | Invitado |
| Día: | jueves, 3 de abril de 2025, 16:48 |
1. ¿Cómo se replica el ADN?
Observa el siguiente video introductorio:
Los experimentos mostraron que las cadenas simples de ADN podrían ser replicadas en un tubo de ensayo que contenía sustratos simples y una enzima. Luego, un experimento clásico demostró que cada una de las dos cadenas de la doble hélice puede actuar como molde para una cadena de ADN nueva.
 |
Arthur Kornberg demostró que es posible sintetizar el ADN con la misma composición de bases del ADN parental en un tubo de ensayo que contiene tres sustancias: |
|---|---|
 |
|
 |
|
 |
|
Replicación en la célula:
Ocurre en varios pasos:
- La doble hélice del ADN se desenrolla hasta separar las dos cadenas molde y las deja disponibles para el nuevo apareamiento de bases. Participa una enzima denominada ADN helicasa.
- Se unen dos nucleósidos mediante enlaces fosfodiéster a cada cadena en crecimiento en una secuencia determinada por el apareamiento de bases complementarias con las bases de la cadena doble. Las ADN polimerasas agregan nucleósidos a la cadena en crecimiento, se necesita una cadena iniciadora llamada cebador.
- Las dos cadenas de ADN crecen de forma diferente. Una cadena en replicación creciente (la cadena o hebra conductora) apunta en la dirección "correcta" para crecer de manera continua en su extremo 3´a medida que la horquilla se abre. La otra cadena nueva (la cadena o hebra retrasada) apunta en la dirección "errónea": a medida que la horquilla se abre más adelante, se expone el extremo 3´y se aleja cada vez más de la horquilla de replicación. La síntesis de la hebra retrasada requiere trabajar en tramos relativamente pequeños y discontinuos, estos tramos se denominan fragmentos de Okasaki. La enzima ligasa cataliza la formación de los enlaces de unión de los fragmentos en una sola cadena.
1.1. Aplicación 1: PCR
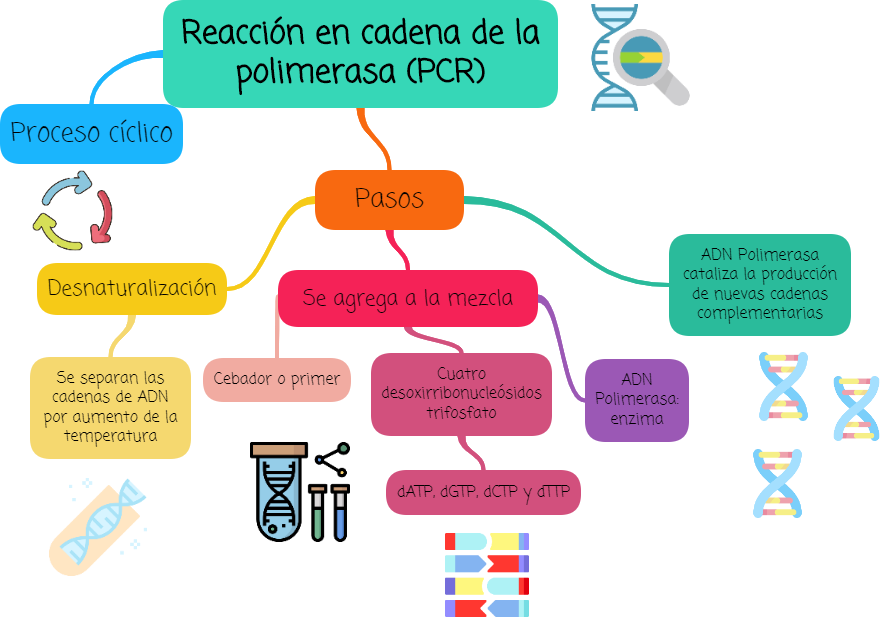
La reacción en cadena de la polimerasa hace múltiples copias del ADN:
Puesto que el ADN puede ser replicado en el laboratorio, es posible hacer copias de una secuencia de ADN. La técnica de reacción en cadena de la polimerasa (PCR) esencialmente automatiza este proceso al copiar una región corta de ADN muchas veces en un tubo de ensayo.
La PCR es un proceso cíclico en el cual una secuencia de pasos que se repite una y otra vez:
- Se separan los fragmentos de ADN en una cadena simple mediante calentamiento (desnaturalización).
- Se agrega a la mezcla un cebador corto (también llamado primer) sintetizado artificialmente, con los cuatro desoxirribonucleósidos trifosfato (dATP, dGTP, dCTP y dTTP) y la ADN polimerasa.
- La ADN polimerasa cataliza la producción de nuevas cadenas complementarias.
Un solo ciclo toma unos minutos en duplicar la cantidad de ADN, la repetición del ciclo muchas veces conduce a un incremento exponencial en el número de copias de la secuencia de ADN, este proceso se conoce como amplificación de la secuencia.
Un problema inicial con la PCR fue sus requerimientos de temperatura. Para desnaturalizar el ADN la temperatura debe alcanzar los 90 °C, una temperatura que destruye la mayoría de las ADN polimerasas. La técnica no sería práctica si se tuviera que añadir una nueva polimerasa después de cada ciclo de desnaturalización.
 |
Este problema fue resuelto por la naturaleza. En las aguas termales del Parque Nacional de Yellowstone, como también otros lugares de temperaturas altas, vive una bacteria denominada Thermus aquaticus. Thomas Brock y sus colegas investigaron la manera por la cual este organismo sobrevive a temperaturas de hasta 95 °C. Descubrieron que esta bacteria tiene una ADN polimerasa que no se desnaturaliza a esas altas temperaturas. ¿Por qué no utilizar esta polimerasa en la PCR? La idea funcionó y el bioquímico Kary Mullis recibió por ella el premio Nobel. |
|---|
En resumen:
1.2. Aplicación 2: Método de Sanger
Se puede determinar la secuencia nucleotídica del ADN:
 |
En 1977, Frederick Sanger desarrolló el método de secuenciación de ADN conocido como método de Sanger. Dos años más tarde empleó esta técnica para secuenciar el genoma del bacteriófago Phi-X174, el primer ácido nucleico secuenciado totalmente en la historia. Realizó este trabajo manualmente, sin ayuda de ningún automatismo. Este trabajo fue base fundamental para proyectos tan ambiciosos como el Proyecto Genoma Humano y por él se le concedió su segundo Premio Nobel en 1980, que compartió con Walter Gilbert. |
|---|
Esta técnica se basa en el uso de nucleósidos alterados artificialmente. Si la desoxirribosa se reemplaza con 2,3-didesoxirribosa, el didesoxirribonucleósido trifosfato (ddNTP) resultante será añadido por la ADN polimerasa a una cadena de polinucleotídica en crecimiento. Sin embargo, como los ddNTP carecen de un grupo (-OH) en la posición 3´, no se podrá añadir el próximo nucleótido. En consecuencia, la síntesis se detiene en la posición en que se ha incorporado el ddNTP en el extremo en crecimiento de una cadena de ADN.
Para determinar la secuencia de ADN, se desnaturaliza un fragmento (por lo general, de no más de 700 pares de bases de longitud). Las cadenas simples se colocan en un tubo de ensayo y se mezclan con:
- ADN polimerasa, para sintetizar la cadena complementaria.
- cebadores cortos sintetizados artificialmente, apropiados para la secuencia de ADN.
- los cuatro dNTP (dATP, dGTP, dCTP y dGTP).
- cantidades pequeñas de los cuatro ddNTP, cada uno unido a un "marcador" fluorescente que emite un color de luz diferente.
Conjunto de Fichas creado con GoConqr por Anarella Gatto
La replicación avanza y pronto el tubo de ensayo contiene una mezcla de cadenas de ADN molde y, en breve, cadenas complementarias nuevas. Las cadenas nuevas, cada una de las cuales finaliza con un ddNTP fluorescente, son de longitudes variables. Por ejemplo, cada vez que se llega a una T en la cadena molde, la ADN polimerasa añade, ya sea un dATP o un ddATP, a la cadena complementaria en crecimiento. Si se añade un dATP, la cadena continúa creciendo. Si se añade un ddATP, el crecimiento se detiene.
Después de que se ha permitido proseguir la replicación por un rato, se desnaturalizan los nuevos fragmentos de ADN de sus moldes y se los somete a electroforesis. Esta técnica separa los fragmentos de ADN por longitud y puede detectar diferencias en la longitud de los fragmentos tan cortos como una base. Durante la corrida electroforética, los fragmentos pasan en función de la longitud creciente a través de un rayo láser que excita los marcadores fluorescentes. Luego se detecta la luz emitida.
La información resultante -es decir, qué color de fluorescencia y, por lo tanto, cuál ddNTP está en el extremo de la cadena de esa longitud- se ingresa en una computadora. Este procesa la información e imprime la secuencia de ADN del fragmento. La secuenciación del ADN ha formado las bases de la nueva ciencia de la genómica.
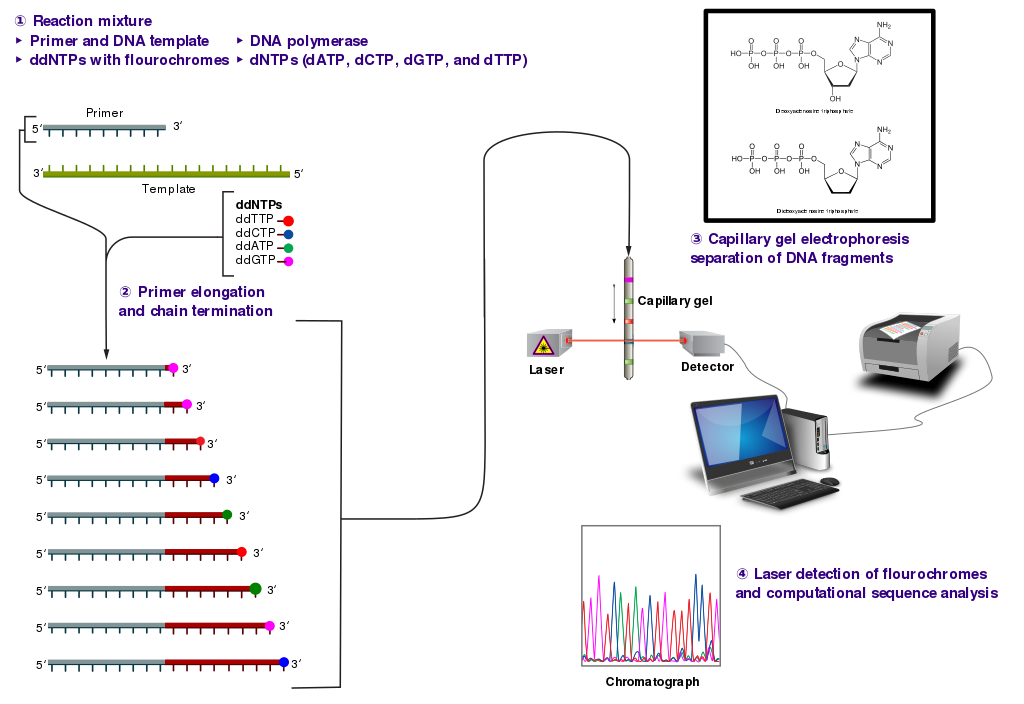
El diagrama anterior representa los pasos seguidos en la secuenciación.
2. La transcripción de la información (del ADN al ARN)
El funcionamiento de una célula depende en gran medida de las proteínas. Estas cumplen un papel fundamental para los seres vivos, ya sea como componentes estructurales, o bien como sustancias de reserva. También pertenecen al grupo de las proteínas las enzimas, moléculas de acción catalítica, y los anticuerpos, moléculas que participan en la defensa del organismo.
Como ya hemos visto las proteínas están formadas por 20 aminoácidos diferentes, la unión de varios aminoácidos forma una cadena peptídica que se caracteriza no solo por el número y el tipo de aminoácidos que la componen, sino que también por la secuencia en que se encuentran. De dicha estructura dependerá la configuración espacial de la molécula y su función.
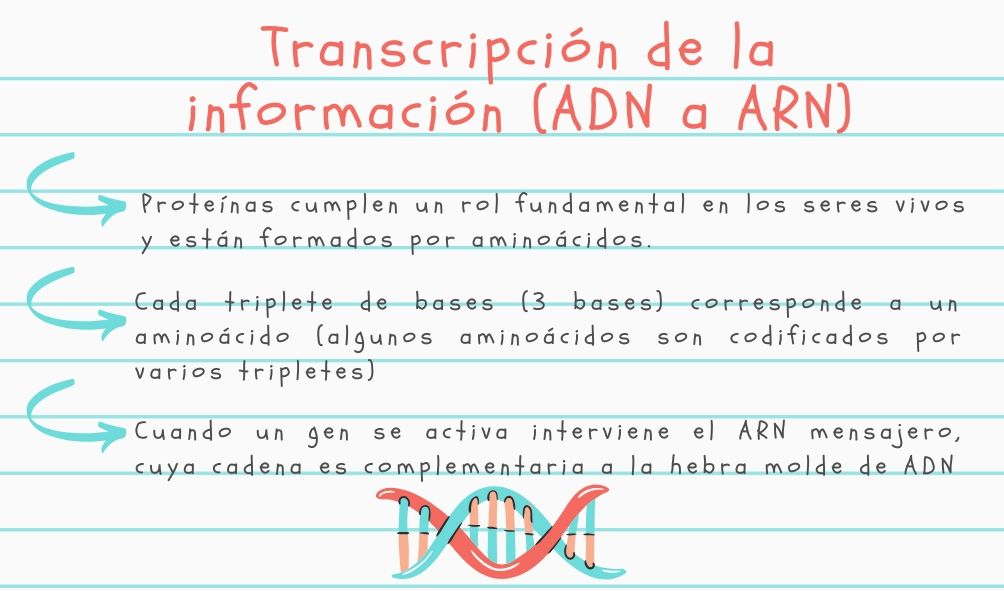
¿Cómo puede un segmento de ADN determinar la secuencia de una proteína? El código es simple, a cada triplete de bases corresponde a un aminoácido (en algunos casos hay varios tripletes que se corresponden a un mismo aminoácido como se analizará en el siguiente capítulo). Cambios en la secuencia de bases del ADN pueden tener como consecuencia la sustitución de un aminoácido por otro.
Cuando un gen se activa, la información no pasa directamente del ADN a los aminoácidos, siendo necesaria la intervención de un intermediario, que es el ARN mensajero (ARNm). Obsérvese que, además de la diferencia citada en relación al glúcidos, el ARNm es una molécula de única cadena y de menor longitud que el ADN.
El ARNm es sintetizado como una cadena complementaria de una de las hebras (hebra
molde), de modo que en la secuencia la única diferencia con la hebra codificante es la
sustitución de timina por uracilo. Una vez transcrita la información, el ARNm la lleva al
citoplasma para el encuentro con la maquinaria celular responsable del montaje del
péptido.
En resumen:
Observa el siguiente video:
3. La traducción de la información (del ARNm a proteína)
La traducción del lenguaje de los ácidos nucleicos al lenguaje de las proteínas permite el montaje de la cadena de aminoácidos en un cierto orden. De este modo, se establece en la célula un flujo de información genética que sigue una dirección única: del ADN al ARN, del ARN al péptido.
Una excepción a esta regla son los retrovirus, cuyo material hereditario es el ARN, porque
cuentan con una enzima (transcriptasa reversa) que les permite transcribir la información
en sentido ARN-ADN.
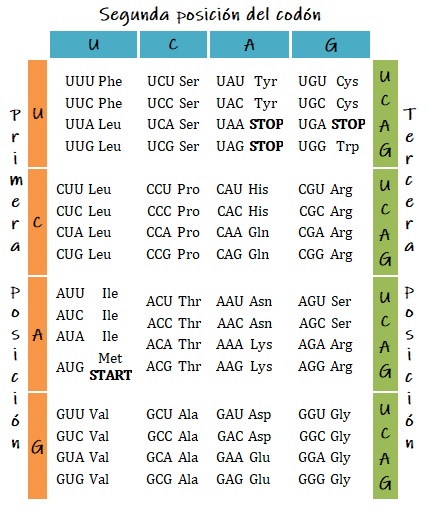
La tabla nos muestra cuáles aminoácidos se corresponden con los diferentes codones o tripletes de bases de ARNm. Algunos son codificados por un único triplete, como el triptófano (UGG) o la metionina (AUG): otros admiten varios codones que resultan sinónimos, por ejemplo, la prolina (CCU, CCC, CCA, CCG).
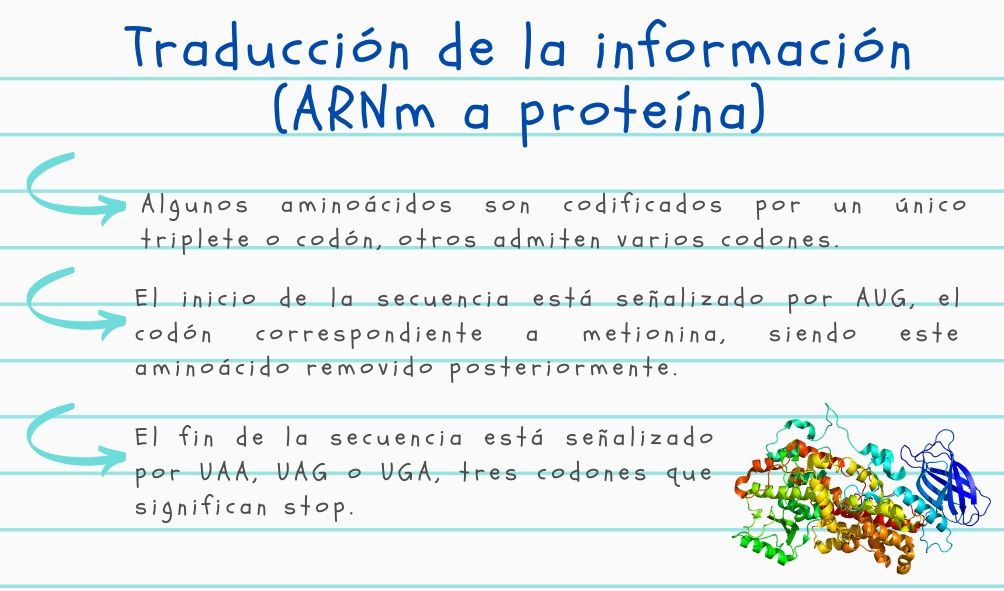
El inicio de la secuencia está señalizado por AUG, el codón correspondiente a metionina, siendo este aminoácido removido posteriormente. El fin de la secuencia está señalizado por UAA, UAG o UGA, tres codones que significan stop, así como en nuestro lenguaje el punto representa el fin de una frase.
Los cambios en la secuencia de bases del ADN pueden tener como consecuencia la sustitución de un aminoácido por otro. En la beta globina humana, si GUG es sustituido por CGU, el aminoácido valina será sustituido por leucina. Pero en función de la sinonimia del código, si el triplete GUG fuera sustituido por GUA o GUC, el aminoácido codificado seguirá siendo valina. Pérdidas o adiciones de una base modifican el resto de la secuencia del péptido.
Las mutaciones puntuales corresponden a pequeños cambios de la secuencia, debidas a errores en la duplicación del ADN. Su frecuencia aumenta en presencia de algunos agentes químicos y físicos como la luz ultravioleta y los rayos X.
En resumen:
Observa el siguiente video:
3.1. Sabías que...
¿Puede un gen codificar varios polipéptidos? Contrariamente a la visión tradicional que consideraba al gen como una secuencia de ADN codificante de un único polipéptido, hoy en día sabemos que la gran mayoría de los genes humanos puede codificar varios polipéptidos. Los mecanismos nucleares de corte y re-unión del transcrito pueden originar diversos ARNm que son traducidos como péptidos diferentes. Esto explicaría por qué 20.000 a 25.000 genes serían suficientes para codificar un ser humano. |
 |
|---|---|
¿Qué porcentaje del ADN codifica proteínas? Las secuencias codificantes de proteínas corresponden, aproximadamente, a 1,5 % del genoma nuclear. En el resto del ADN, mal llamado “basura”, se encuentran diversos tipos de secuencias cuya función está siendo estudiada. |
 |
4. Créditos
Autoría de los materiales utilizados: Profesora Anarella Gatto.
Esta obra está bajo una Licencia Creative Commons Atribución 4.0 Internacional.
Bibliografía consultada:
- Sadava, D., Heller, G., Orians, G., Purves, W. y Hillis, D. (2009). Vida. La Ciencia de la Biología. (8va edición). Buenos Aires, Argentina: Panamericana.
- Nelson, D. y Cox, M. (2005). Lehninger Principios de Bioquímica. (Cuarta edición). Omega.
- Malajovich, M. (s.f.). ADN, ARN E INFORMACIÓN. Guía de actividades. Biotecnología: enseñanza y divulgación.
- UdelaR. (s.f.). Código genético. Recuperado de: http://uvigen.fcien.edu.uy/utem/Infgen/infocod.html
- Alegría, Mónica y otros. (1999). Química II. Argentina: Santillana.
- Método de Sanger. (s.f.). En Wikipedia. Recuperado el 28 de mayo de 2020 de: https://es.wikipedia.org/wiki/M%C3%A9todo_de_Sanger
Videos, páginas y/o simuladores utilizados:
- Educar Portal. (2020, marzo 3). Duplicación del ADN. [Archivo de video]. Recuperado de: https://youtu.be/x2WmCYqjOD4.
- Educar Portal. (2020, marzo 10). Transcripción del ADN. [Archivo de video]. Recuperado de: https://youtu.be/k2I04EIqL9M.
- Educar Portal. (2020, marzo 10). Traducción del ADN. [Archivo de video]. Recuperado de: https://youtu.be/qGk1dfa9wOQ.
Las imágenes utilizadas fueron tomadas de:
- Secuenciación Sanger. https://commons.wikimedia.org/wiki/File:Sanger-sequencing.svg
- Frederick Sanger. https://es.wikipedia.org/wiki/Frederick_Sanger#/media/Archivo:Frederick_Sanger2.jpg
- Kary Mullis. https://es.wikipedia.org/wiki/Kary_Mullis#/media/Archivo:Kary_Mullis.jpg
- Arthur Kornberg. https://es.m.wikipedia.org/wiki/Archivo:Arthur_Kornberg.jpg
- Polimerasa. https://es.wikipedia.org/wiki/Polimerasa#/media/Archivo:Taq_polimerase.png
- Íconos de Flaticon:
- https://image.flaticon.com/icons/svg/2885/2885106.svg
- https://image.flaticon.com/icons/svg/1809/1809162.svg
- https://image.flaticon.com/icons/svg/1284/1284088.svg
- https://image.flaticon.com/icons/svg/1087/1087538.svg
- https://image.flaticon.com/icons/svg/1087/1087523.svg
- https://image.flaticon.com/icons/png/512/2547/2547769.png
- https://image.flaticon.com/icons/svg/1308/1308858.svg
- https://image.flaticon.com/icons/svg/1308/1308876.svg
- https://image.flaticon.com/icons/svg/2372/2372773.svg