Del ácido desoxirribonucleico (ADN) a las proteínas
1. ¿Cómo se replica el ADN?
1.2. Aplicación 2: Método de Sanger
Se puede determinar la secuencia nucleotídica del ADN:
 |
En 1977, Frederick Sanger desarrolló el método de secuenciación de ADN conocido como método de Sanger. Dos años más tarde empleó esta técnica para secuenciar el genoma del bacteriófago Phi-X174, el primer ácido nucleico secuenciado totalmente en la historia. Realizó este trabajo manualmente, sin ayuda de ningún automatismo. Este trabajo fue base fundamental para proyectos tan ambiciosos como el Proyecto Genoma Humano y por él se le concedió su segundo Premio Nobel en 1980, que compartió con Walter Gilbert. |
|---|
Esta técnica se basa en el uso de nucleósidos alterados artificialmente. Si la desoxirribosa se reemplaza con 2,3-didesoxirribosa, el didesoxirribonucleósido trifosfato (ddNTP) resultante será añadido por la ADN polimerasa a una cadena de polinucleotídica en crecimiento. Sin embargo, como los ddNTP carecen de un grupo (-OH) en la posición 3´, no se podrá añadir el próximo nucleótido. En consecuencia, la síntesis se detiene en la posición en que se ha incorporado el ddNTP en el extremo en crecimiento de una cadena de ADN.
Para determinar la secuencia de ADN, se desnaturaliza un fragmento (por lo general, de no más de 700 pares de bases de longitud). Las cadenas simples se colocan en un tubo de ensayo y se mezclan con:
- ADN polimerasa, para sintetizar la cadena complementaria.
- cebadores cortos sintetizados artificialmente, apropiados para la secuencia de ADN.
- los cuatro dNTP (dATP, dGTP, dCTP y dGTP).
- cantidades pequeñas de los cuatro ddNTP, cada uno unido a un "marcador" fluorescente que emite un color de luz diferente.
Conjunto de Fichas creado con GoConqr por Anarella Gatto
La replicación avanza y pronto el tubo de ensayo contiene una mezcla de cadenas de ADN molde y, en breve, cadenas complementarias nuevas. Las cadenas nuevas, cada una de las cuales finaliza con un ddNTP fluorescente, son de longitudes variables. Por ejemplo, cada vez que se llega a una T en la cadena molde, la ADN polimerasa añade, ya sea un dATP o un ddATP, a la cadena complementaria en crecimiento. Si se añade un dATP, la cadena continúa creciendo. Si se añade un ddATP, el crecimiento se detiene.
Después de que se ha permitido proseguir la replicación por un rato, se desnaturalizan los nuevos fragmentos de ADN de sus moldes y se los somete a electroforesis. Esta técnica separa los fragmentos de ADN por longitud y puede detectar diferencias en la longitud de los fragmentos tan cortos como una base. Durante la corrida electroforética, los fragmentos pasan en función de la longitud creciente a través de un rayo láser que excita los marcadores fluorescentes. Luego se detecta la luz emitida.
La información resultante -es decir, qué color de fluorescencia y, por lo tanto, cuál ddNTP está en el extremo de la cadena de esa longitud- se ingresa en una computadora. Este procesa la información e imprime la secuencia de ADN del fragmento. La secuenciación del ADN ha formado las bases de la nueva ciencia de la genómica.
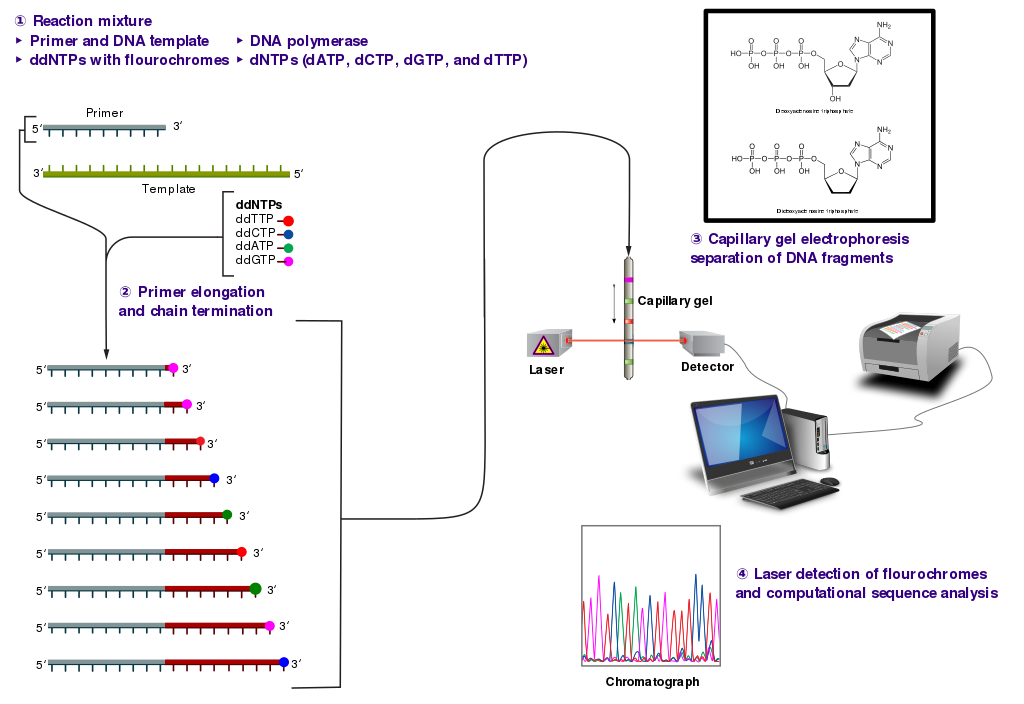
El diagrama anterior representa los pasos seguidos en la secuenciación.